Python OpenCV/scikit-image
Verfasst: Donnerstag 15. April 2021, 13:38
Huhu,
und zwar habe ich gerade folgendes Problem, um Informationen aus Rezepten zuverlässiger zu extrahieren müsste ich bestmöglich den kompletten Hintergrund entfernen, damit nur noch die gedruckte (schwarz/graue) Schrift über bleibt.
Das funktioniert mit dem aktuellen Code auch gut wenn die Schrift wirklich schwarz gedruckt ist, allerdings ist das eher selten der Fall meistens ist der Druck blass und hellgrau.
Somit wird dementsprechend auch die Schrift teilweise entfernt bzw. gebrochen (was mit erode und dilate auch nicht zu beheben ist).
Ein Originalbild kann ich aus Datenschutzgründen leider nicht zeigen, allerdings kann man sich den Anwendungsfall denke ganz gut vorstellen
Gibt es eine simple Möglichkeit den Hintergrund zu entfernen bzw. alle Pixel welche einen höheren Rotanteil als x besitzen "einfach" auf weiß 255 zu setzen ?
Oder vlt. eine ganz andere Idee / Ansatz ?
Eine weitere Idee war es einen Autoencoder dafür zu verwenden/trainieren (allerdings wäre das der letzte Schritt/Versuch) bevorzugt sind CV Lösungen.
Ein weiterer erfolgloser Versuch war: cv2.absdiff(reales_img, diese_blanko_vorlage)

bisheriger Code:
Anhang:
Zur Verdeutlichung 2 schlechte Ausschnitte und 1 gutes:
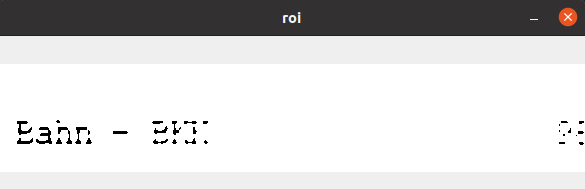


Danke euch
LG Felix
und zwar habe ich gerade folgendes Problem, um Informationen aus Rezepten zuverlässiger zu extrahieren müsste ich bestmöglich den kompletten Hintergrund entfernen, damit nur noch die gedruckte (schwarz/graue) Schrift über bleibt.
Das funktioniert mit dem aktuellen Code auch gut wenn die Schrift wirklich schwarz gedruckt ist, allerdings ist das eher selten der Fall meistens ist der Druck blass und hellgrau.
Somit wird dementsprechend auch die Schrift teilweise entfernt bzw. gebrochen (was mit erode und dilate auch nicht zu beheben ist).
Ein Originalbild kann ich aus Datenschutzgründen leider nicht zeigen, allerdings kann man sich den Anwendungsfall denke ganz gut vorstellen
Gibt es eine simple Möglichkeit den Hintergrund zu entfernen bzw. alle Pixel welche einen höheren Rotanteil als x besitzen "einfach" auf weiß 255 zu setzen ?
Oder vlt. eine ganz andere Idee / Ansatz ?
Eine weitere Idee war es einen Autoencoder dafür zu verwenden/trainieren (allerdings wäre das der letzte Schritt/Versuch) bevorzugt sind CV Lösungen.
Ein weiterer erfolgloser Versuch war: cv2.absdiff(reales_img, diese_blanko_vorlage)

bisheriger Code:
Code: Alles auswählen
img = resize_and_rotate_image(path, res_height=900, res_width=1200)
value = check_signed(img)
result_dict['recipe']['isSigned']['value'] = value
if value:
print('have detect signature in prescription')
else: print('no signature found')
img = cv2.GaussianBlur(img,(3,3),2)
kernel = cv2.getStructuringElement(cv2.MORPH_RECT, (2,2))
morph_open = cv2.morphologyEx(img, cv2.MORPH_OPEN, kernel)
thresh = cv2.threshold(morph_open,210,255,cv2.THRESH_BINARY)[1]
mask = cv2.inRange(thresh, 0, 120)
kernel = np.array([[-1,-1,-1],
[-1, 9,-1],
[-1,-1,-1]])
sharpened = cv2.filter2D(mask, -1, kernel)
morph_close = cv2.morphologyEx(sharpened, cv2.MORPH_CLOSE, kernel)
img = cv2.bitwise_not(morph_close)Zur Verdeutlichung 2 schlechte Ausschnitte und 1 gutes:



Danke euch
LG Felix

